
Plotear el mapa de ocurrencia de la especie que desees nunca fue más sencillo. Gracias al API de GBIF (Global Biodiversity Information Facility), nos permite sacarle el máximo provecho a esa vasta base de datos de biodiversidad. ¿Quieres aprender cómo?
Preprocesando la data
# Instalar los paquetes
install.packages("rgbif")
install.packages("maptools")
install.packages("rgrass7")
install.packages("raster")
install.packages("mapr")
install.packages("rgdal")
# Activar los paquetes
# el paquete sp se instala como
# dependiente de rgbif
library(rgbif)
library(maptools)
library(rgrass7)
library(raster)
library(sp)
library(mapr)
library(rgdal)
1. Definir la extensión del mapa
En este ejemplo trabajaré con el noroeste de Perú. Debemos ingresar las coordenadas decimales sexagesimales en el orden este, oeste, sur y norte. Para ello, les recomiendo ingresar a esta web, que en realidad sirve para bajar mapas DEM (digital elevation model) de todo el mundo, pero podemos emplearla para dibujar un polígono en el mapa y copiar las coordenadas de su extensión, como se muestra en la imagen, para luego reemplazar las que les coloco aquí en el código.
extend <- c(-81.984694500359, -76.1835027785539, -10.943478993008, -3.1601456596749777)
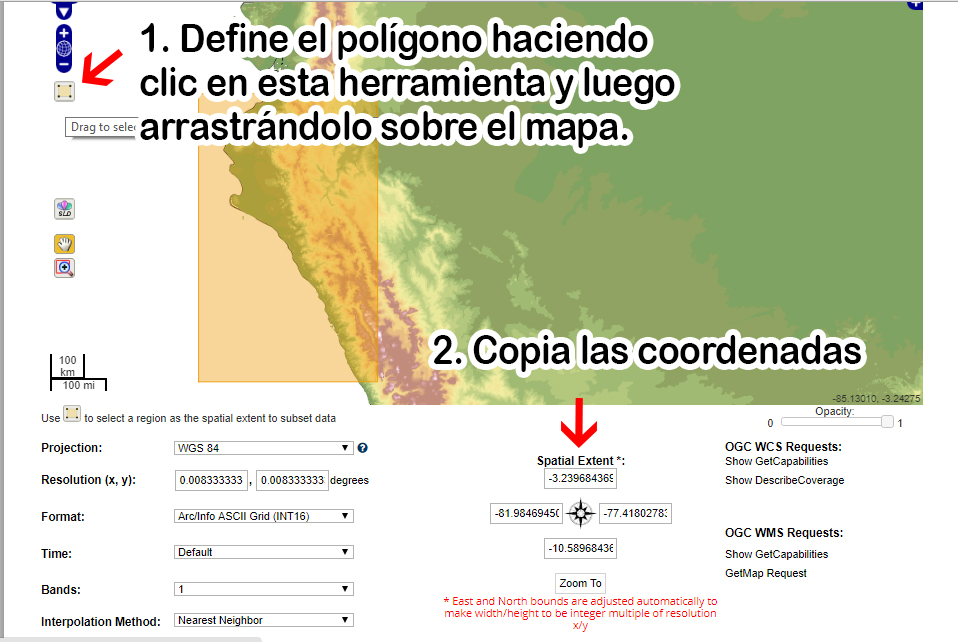
Corroboramos en R la ubicación del polígono en el mapa del mundo cargado en los paquetes corriendo el siguiente código:
# Plotear el mapa del mundo
data(wrld_simpl)
plot(wrld_simpl, col = "white", axes = T)
# Ploteamos el polígono que definimos
# encima del mapas del mundo
area <- as(extent(NCext), "SpatialPolygons")
plot(area, col = "red", add=TRUE)
2. Crear el Shapefile de la extensión en QGIS
En QGIS, abre la capa shapefile (.shp) del mapa que deseas emplear y recorta el área de la extensión definida anteriormente en la web. En ella proyectaremos los puntos de ocurrencia del GBIF.
- Abrimos el QGIS
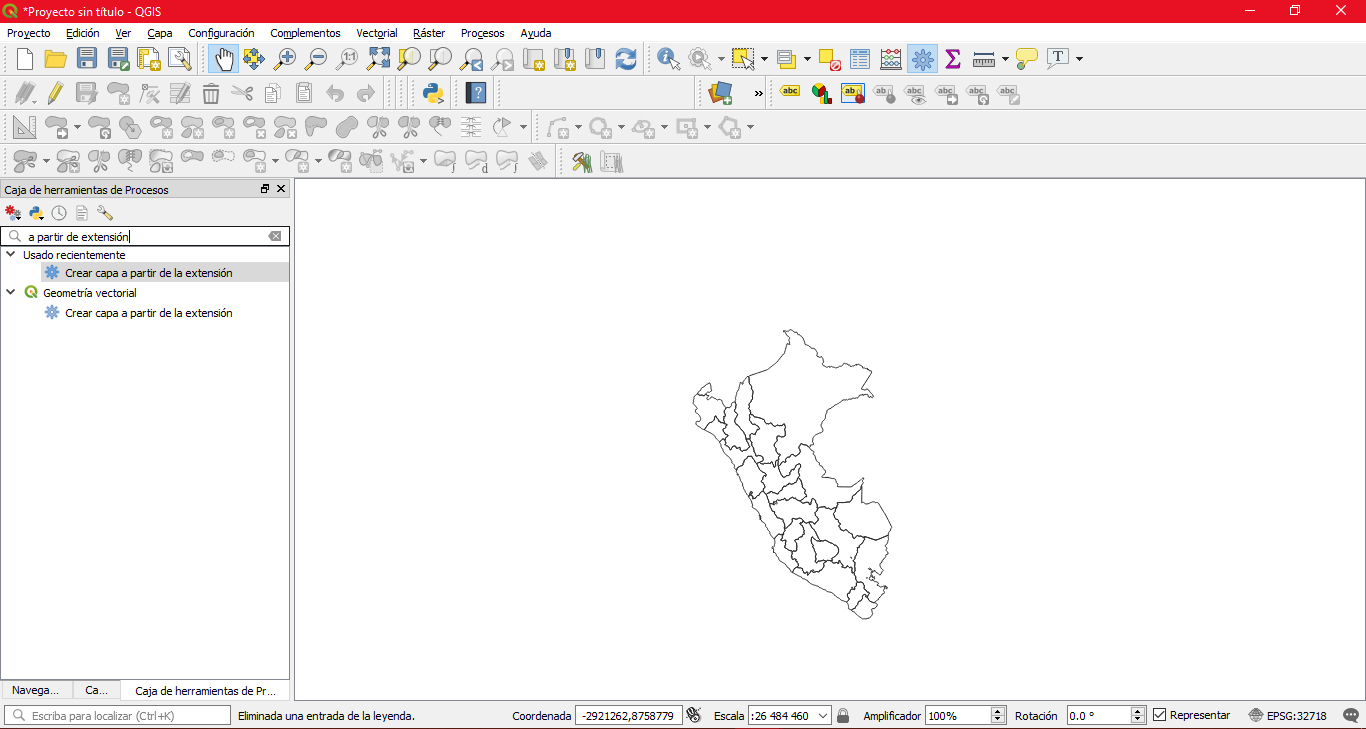
- Abrimos la capa (geopolítica) de donde queremos extraer el área de estudios.
- Buscamos la herramienta «Crear capa a partir de extensión» en la Caja de Herramientas de Procesos de QGIS.
- En la casilla «extensión» deberán colocar la extensión definida en extend y adicionar el sistema de coordenadas WGS84 entre corchetes, que es en el que se tomaron los datos en la web, de la siguiente manera:
-81.984694500359, -76.1835027785539, -10.943478993008, -3.1601456596749777 [EPSG:4326] - Aceptar y cerrar.
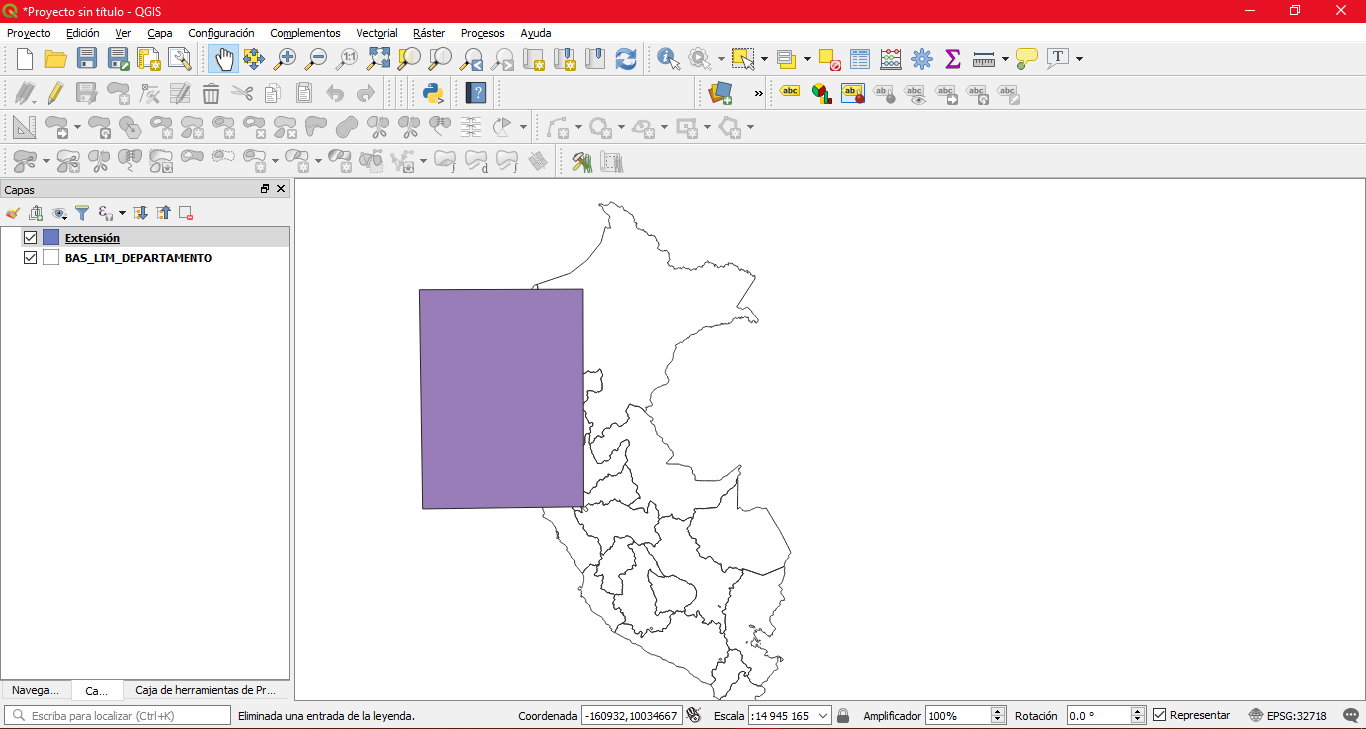
- Ahora, cortaremos el mapa original en base a la extensión del polígono con el geoproceso vectorial «cortar».
- En la casilla «Capa de entrada» seleccionamos lo que queremos cortar, es decir, el mapa geopolítico original.
- En la casilla «Capa de corte», el molde de corte, el polígono.
- Guardamos en nuestros archivos con un nombre simple, por ejemplo «no_peru.shp».
- Queda todo listo para continuar en R.
NOTA: Debemos asegurarnos siempre que las capas que usemos, así como el polígono geopolítico final que exportemos, deben de estar proyectadas en WGS84 (código QGIS: 4326). Sino tendremos problemas al proyectar los datos de GBIF en nuestro mapa. Si tus capas no están en WGS84, usa la herramienta reproyectar de QGIS.
Crear mapas de ocurrencia
Esta es la versión simple del mapa. Siempre podremos recurrir más herramientas de las paqueterías de R para hacer que nuestro mapa luzca mejor, más profesional. En este primer ejemplo veremos el resultado crudo del proceso. En el ejemplo utilicé los datos de una especie de ave endémica de Perú, Phytotoma raimondii.
# Importamos el shapefile de nuestro
# polígono geopolítico recortado
map = readOGR("C:/Archivos/SIG/Projects/16 Mapa de aves libro/mapa/noperu.shp")
# Descargamos los datos de presencia de GBIF.
# Es una buena práctica nombrar al archivo con las iniciales de la especie.
# define la especie en scientificName =""
Phyra <- occ_data(scientificName="Phytotoma raimondii", hasCoordinate=TRUE, geometry=NCext,
hasGeospatialIssue=FALSE)
map_ggplot(Phyra, map=map)

La función map_ggplot tiene precargados los mapas de el mundo, Estados Unidos, Francia y Nueva Zelanda. Si tuvieran el mapa de cada país nos habríamos ahorrado todo el preprocesamiento en QGIS. Por ejemplo, llamando el argumento map=»world», obtendremos:

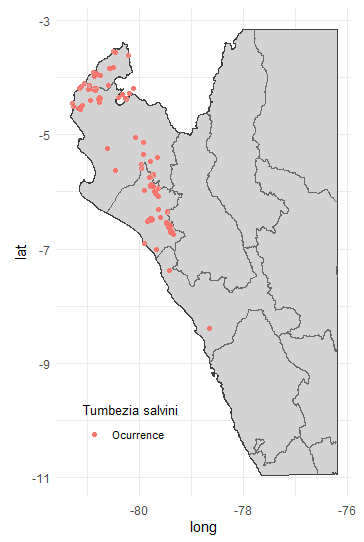
Para mapas con mejor calidad utiliza el código en ggplot2. Utilicemos ahora los datos de la especie de ave casi amenazada quasi-endémica de Perú Tumbezia salvini:
ggextension = map
b <- ggplot(ggextension, aes(long, lat)) + geom_polygon() + coord_equal()
b <- ggplot() + coord_fixed(1.1)
b <- b + geom_polygon(data = area, aes(x=long, y = lat, group = group),
fill="white", col="grey", lwd=0.3)
b <- b + geom_point(data=TS$data, aes(x = decimalLongitude, y=decimalLatitude,
color="Ocurrence"), cex=1.5) + labs(color = "Tumbezia salvini")
b <- b + geom_polygon(data = area, aes(x=long, y = lat, group = group),
fill=rgb(1,1,1,0), col=rgb(0.8,0.8,0.8,0.3), lwd=0.1)
b <- b + theme(legend.position=c(0.25,0.15),
legend.title = element_text(color = "black", size = 10),
legend.text = element_text(color = "black", size=8))
b
NOTA: No se olviden de modificar los valores de ggextension, area, color (con el nombre de la especie), y data source de GBIF que en este ejemplo fue la variable TS. Miren siempre esos detalles en su código.





Deja un comentario